基因Rgls位点的精细定位及分子标记可靠性验证
基因Rgls位点的精细定位及分子标记可靠性验证
高分辨熔解曲线 (High Resolution Melting,HRM) 分析技术是近年来国际上兴起的一种最新的应用于基因突变检测和 SNP 分析的方法。因为所用的荧光染料只能嵌入并结合到双链 DNA 上,因此利用实时 PCR 技术,就能通过实时检测双链DNA 熔解过程中荧光信号值的变化,将 PCR 产物中存在的差异以不同形状熔解曲线的方式直观地展示出来,并且可以借助于专业的分析软件对测试群体实现基于不同形状熔解曲线的基因分型或归类 (殷豪等,2011)。
高分辨熔解 (HRM) 曲线分析技术具有三个突出的优势:一是高通量、高灵敏度、高特异性、低成本且不受检测位点的限制;二是操作简单、快捷、节省时间成本;三是闭管操作,无污染且 DNA 不受损伤,熔解分析后还可以进行凝胶电泳或测序分析。在 LightCycler ? 480分析仪上一次可以完成96个或348个样本的分析,从反应开始到数据生成仅需要90~120 min。目前HRM技术在果树的种质鉴别和基因分型研究中已有应用。吴波等 (2012) 引入高分辨率熔解曲线分型技术对柑橘进行SNP 分型;Ganopoulos等 (2013) 利用 HRM 技术对9个甜樱桃基因上的 SNP 位点进行分析,成功的实现了对21 个甜樱桃品种的鉴别,准确率达到 99.9%;Distefano 等 (2013) 第一次将HRM技术应用于对柑橘品种 SNP 及 InDel的检测,并利用21 个 SNP标记实现了对柑橘不同品种的区别。
分子标记辅助选择育种 (marker assisted selection,MAS) 作为一种高效的现代分子育种技术,已被广泛应用于作物品种选育和遗传改良。它是利用与目标基因紧密连锁的分子标记,来鉴定不同个体的基
本试验利用HRM分析技术对上一章节所开发的SNP 和InDel标记进行筛选和验证,以获得与抗炭疽菌叶枯病基因
第一节 材料与方法
一、植物材料
选择青岛农业大学苹果试验基地 (山东省胶州市) 2009 年种植的,经过人工离体接种鉴定的 ‘金冠’ב富士’ 的F
二、抗感DNA池的构建
同第三章。
三、SNP引物的设计
用于引物设计的SNP 和 InDel位点来源于第四章中所筛选出的位
四、高分辨率熔解曲线分析
PCR扩增和高通量熔解曲线分析在 LightCycler ? 480Ⅱ荧光定量PCR 仪 (Roche) 上进行。反应试剂来自 LightCycler ? 480 High Reso-lution Melting Master试剂盒。反应体系为15 μl,内含10 ng/μl的基因组 DNA 1.0 μl,1×Master Mix 7.5 μl,2.0 mmol/L MgCl
PCR扩增程序为95℃预变性10min,然后按95℃变性10min,55℃退火15s,72℃延伸10s的程序进行45个循环。在PCR循环结束后,立即对扩增产物进行HRM检测,程序为:95℃1min,40℃1min,65℃1s,在65℃升温至95℃的过程中,以25次/℃的频率收集荧光信息,最后降温至40℃。高分辨率熔解曲线分析用LightCycler?480的Gene Scanning软件(1.5version)进行。
五、SNP、InDel标记的筛选验证
用18对 SNP 及30对InDel引物在亲本和抗感基因池中筛选,将出现不同分型的引物在分离群体上进行验证,以确定该标记是否与目的基因连锁。
六、基因Rgls位点的精细定位
将筛选的多态性标记在重组个体上的基因型表现与重组个体的抗病表型进行统一分析,对抗炭疽菌叶枯病基因
七、基因Rgls位点区域内的基因分析
将进一步缩小的
八、分子标记准确性鉴定
SSR标记的PCR产物利用3.5%的琼脂糖凝胶电泳进行标记基因型鉴定。SNP 和InDel标记利用HRM技术进行基因分型鉴定。
第二节 结果与分析
一、SNP及InDel标记的筛选
通过对设计的引物进行 BSA 筛选,获得了与
引物名称引物序列序列长度参考碱基突变碱基位置SNP3955R:CCCTTAAAAGCCATGGAAGAGF:GTTCTGCATAAAAACCTCGCA133AGchr15∶3,955,630-3,955,763SNP4236R:GCTTATCATAAAAAGCAAGACCACF:ATCATATAATTGTGTAATTTAGTAGAACA114AGchr15∶4,236,220-4,236,353SNP4257R:GGAGTCATAAGCCACAACGAGF:TCAGCTTTGAAGCATCCAATT145GAchr15∶4,257,141-4,257,286SNP4299R:GGTTATACATAGAGGCACTTAGAGCF:GCACAAAACTTAGATCAAAGATGAG135TCchr15∶4,299,179-4,299,314

表5-1 SNP、InDel引物筛选
引物名称引物序列序列长度参考碱基突变碱基位置SNP4336R:AGTTCGTTCTTTTCCGTTGCTF:GCGGTCCTGATTCAGGTACAG133CGchr15∶4,336,382-4,336,515SNP4432R:CGAGGAGCAAACGATAGTCAGF:ATTGGTCTCCGAATTAGAAGTCC137AGchr15∶4,432,529-4,432,666indel4199R:ATTGTGAAACCTTGATTGGGF:GAGATTATCCTTATTTTGTGGG156AAAGchr15∶4,198,916-4,199,072indel4227R:AGCGTTGCTATGCTTCTAATGF:AAGATGGAAATGGTATGTGAT81TTCchr15∶4,227,569-4,227,650indel4254R:ATAAAGTCACTTCTAGCACAAATAF:CGAAAAACGCTTTACTTAGG119GGCchr15∶4,254,896-4,255,015indel4305R:GTAAACTCATTAAATTATGCTTGF:TGCTTTACTCCGATTCTTC134CCCAchr15∶4,305,342-4,305,476indel4334R:ATACTATGAGGTGAAGGATTTAAF:GTATCTTCTACATTATCTTTCGTG115TTAchr15∶4,334,597-4,334,712
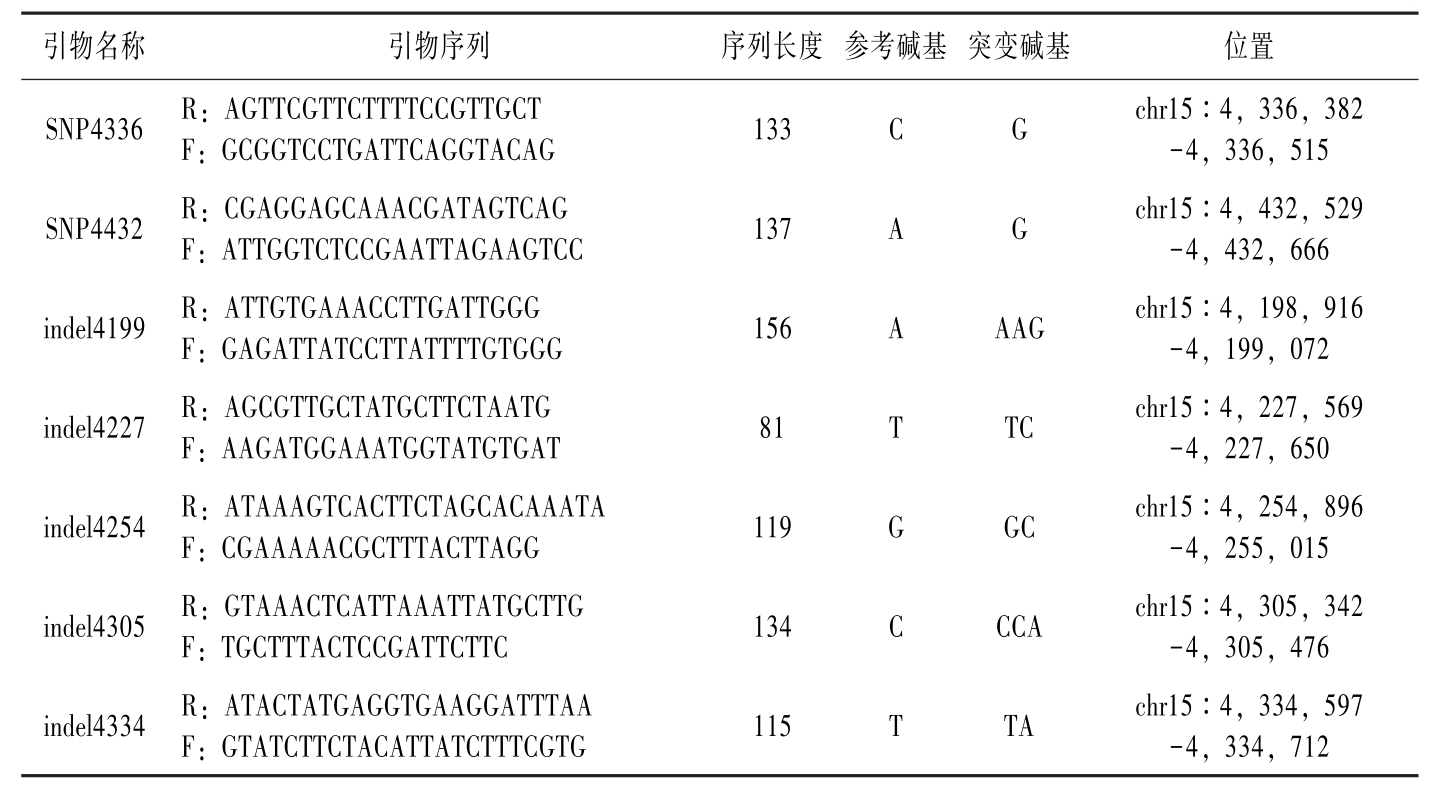
表5-1 SNP、InDel引物筛选(续)-1
二、SNP及InDel标记的验证
将获得的11个标记在207 株 F
三、基因Rgls位点的精细定位
将从全基因组重测序中筛选出并经群体验证的与
四、基因Rgls位点区域内的基因分析
按照基因位置信息从蔷薇科基因组网站 GDR下载基因组15 号染色体4.1~4.3 Mb内基因45个。通过perl脚本整理基因组GFF文件,BLAST同源比对NR数据库,选取最优比对结果。获得目的区段内的45个基因,其中5 个基因功能未知,2 个为转录因子,另外40 个基因均有明确的注释信息 (表5-2)。
序号MDP号码同源基因1MDP0000180944生长素诱导蛋白15A2MDP0000205434非病原性诱导蛋白3MDP0000148158生长素诱导蛋白15A4MDP0000177664蛋白酶体beta亚基5MDP0000177665转录共抑制因子LEUNIG6MDP0000215349转录共抑制因子LEUNIG7MDP0000664885抗烟草花叶病毒蛋白N8MDP0000877582抗烟草花叶病毒蛋白N9MDP0000200748转录因子10MDP0000481972抗烟草花叶病毒蛋白N11MDP0000481973抗烟草花叶病毒蛋白N12MDP0000381897转录因子13MDP0000297052抗烟草花叶病毒蛋白N14MDP0000700563黄瓜素15MDP0000242744阳离子运输调控蛋白216MDP0000551192阳离子运输调控蛋白217MDP0000242745转录因子18MDP0000153007来自转座子TNT1-94的反转录病毒Pol多肽蛋白19MDP0000130036未知功能转录因子20MDP0000199184未知功能转录因子21MDP0000489432三角状五肽链重复包含蛋白22MDP0000318360类LRR丝氨酸/苏氨酸受体蛋白激酶23MDP0000247898类LRR丝氨酸/苏氨酸受体蛋白激酶

表5-2 区段内基因同源比对结果
序号MDP号码同源基因24MDP0000811774核糖核酸酶H2亚基B25MDP0000309446未知功能线粒体蛋白26MDP0000272143单尿苷绑定蛋白1B;与mRNA3’-UTR绑定27MDP0000871880脱水响应蛋白RD2228MDP0000272145酪蛋白激酶Idelta小体29MDP0000167752结构域包含蛋白3430MDP0000167753核糖核酸酶H2亚基B31MDP0000596125核细胞溶解酶TIA-1小体32MDP0000201427拓扑异构酶I33MDP0000149447脱水响应蛋白RD2234MDP0000596128酪蛋白激酶Ideta小体35MDP0000201428UDP-糖转运蛋白36MDP0000201429环核苷酸离子通道蛋白1437MDP0000255274环核苷酸离子通道蛋白1438MDP0000120033丝氨酸精氨酸富集剪接因子。39MDP00001695343-酮脂酰-CoA合酶640MDP0000203647细胞分裂素-O-葡糖基转移酶341MDP0000864010鼠李糖生物合成酶142MDP0000279973肌氨酸氧化酶43MDP0000178030未知蛋白44MDP0000289536肌氨酸氧化酶45MDP0000272940转录因子WRKY11

表5-2 区段内基因同源比对结果(续)-1
为了对基因功能做进一步分析,提取区段内基因的 GO (gene on-tology) 信息,采用基因功能GO分类网站WEBGO (
五、标记在品种(系)中的鉴定
经离体接种鉴定,50 个作为分子标记准确性鉴定材料的品种(系) 中有33 个抗病品种 (系) 和17 个感病品种 (系)。从与抗炭疽菌叶枯病基因紧密连锁的分子标记中挑选出4 个有代表性意义的分子标记SSR标记S0405127,S0304673,SNP 标记 SNP4236,InDel标记InDel4254作为鉴定标记。其中SSR标记 S0405127 与基因
序号品种/系表型S0405127S0304673SNP4236InDel42541海棠RRSRR2斗南RRRRR3福丽RRRRR4福艳RRRRR5红勋1号RRRRS6华帅RRRRR7鲁加1号RRRRR8鲁加2号SSSSS9鲁加4号RRRRR10鲁加6号RRRRR11旭RRRRR12早杂1号RRRRR13威赛克旭RRRRR14五月金RRRRR15早翠绿RRRRR
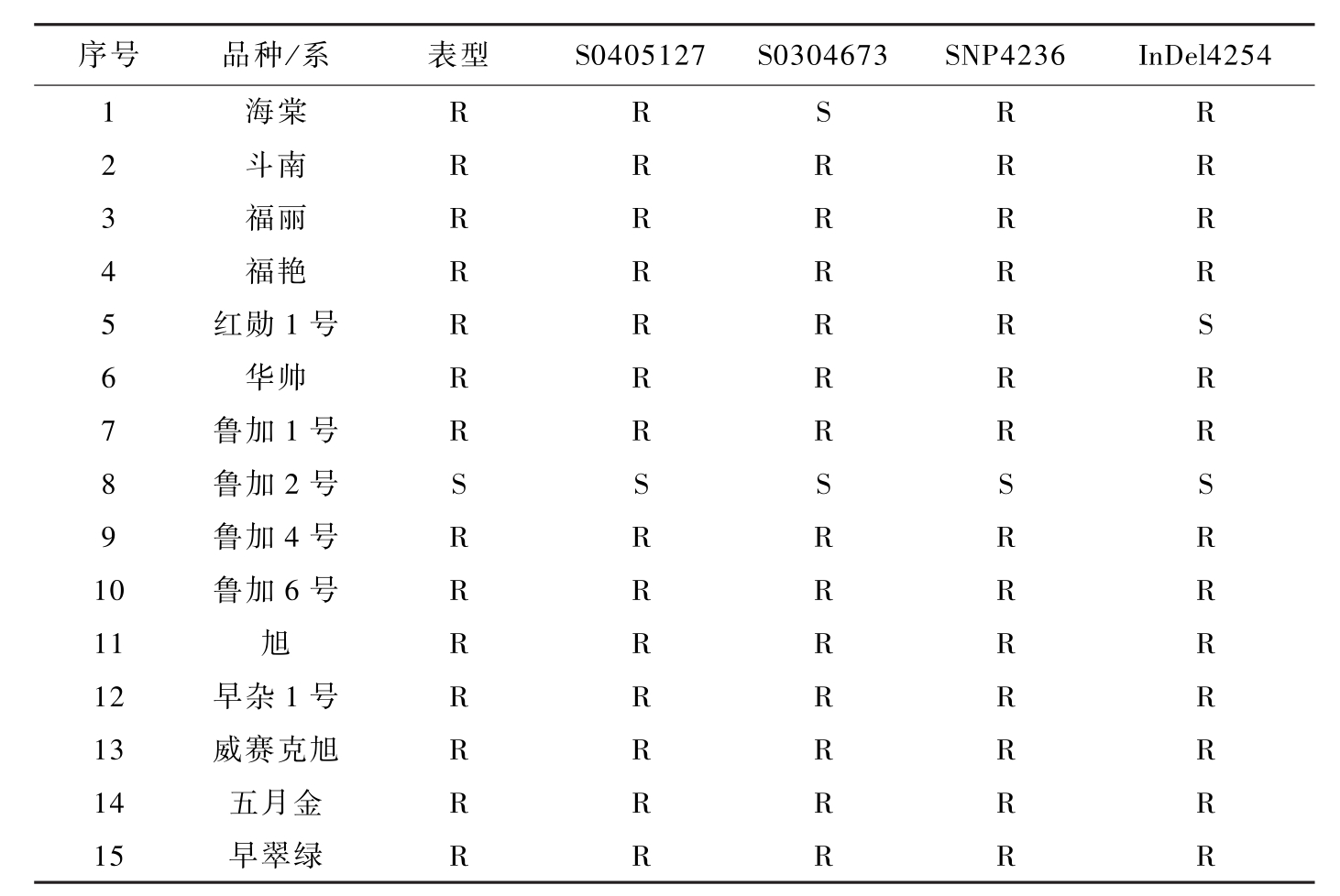
表5-3 四个分子标记对苹果栽培品种和品系的抗病性的鉴定结果
序号品种/系表型S0405127S0304673SNP4236InDel425416国光RRRRR17霞光RRRSS18烟富1RRRRR19福早红SSSSS20嘎拉SSSSS21金冠SSSSS22秦冠SSSSS23华硕RSRRR24瑞丹SSSSS25乙女RRRRR26王林RSRRR27青农红SSSRR28秦冠SSSSS29瑞红SSSSS30赛金RSRRR31红肉1号RRRRR32双阳红SSSSS33望山红RRRRR34新红星RRRRR35青冠RRRRR36弘前富士RRRRR37富士RRRRR387C-102RRRRR39N2SSSSS40PinavaRRRRR417C-35SRRSS4295-161RRRRR4395-231RRRRR4495-232SSSSS4595-32RRRRR4695-93RRRRR477C-104SSSSS487C-105SSSSS497C-106SRRSS507C-107SSSSS
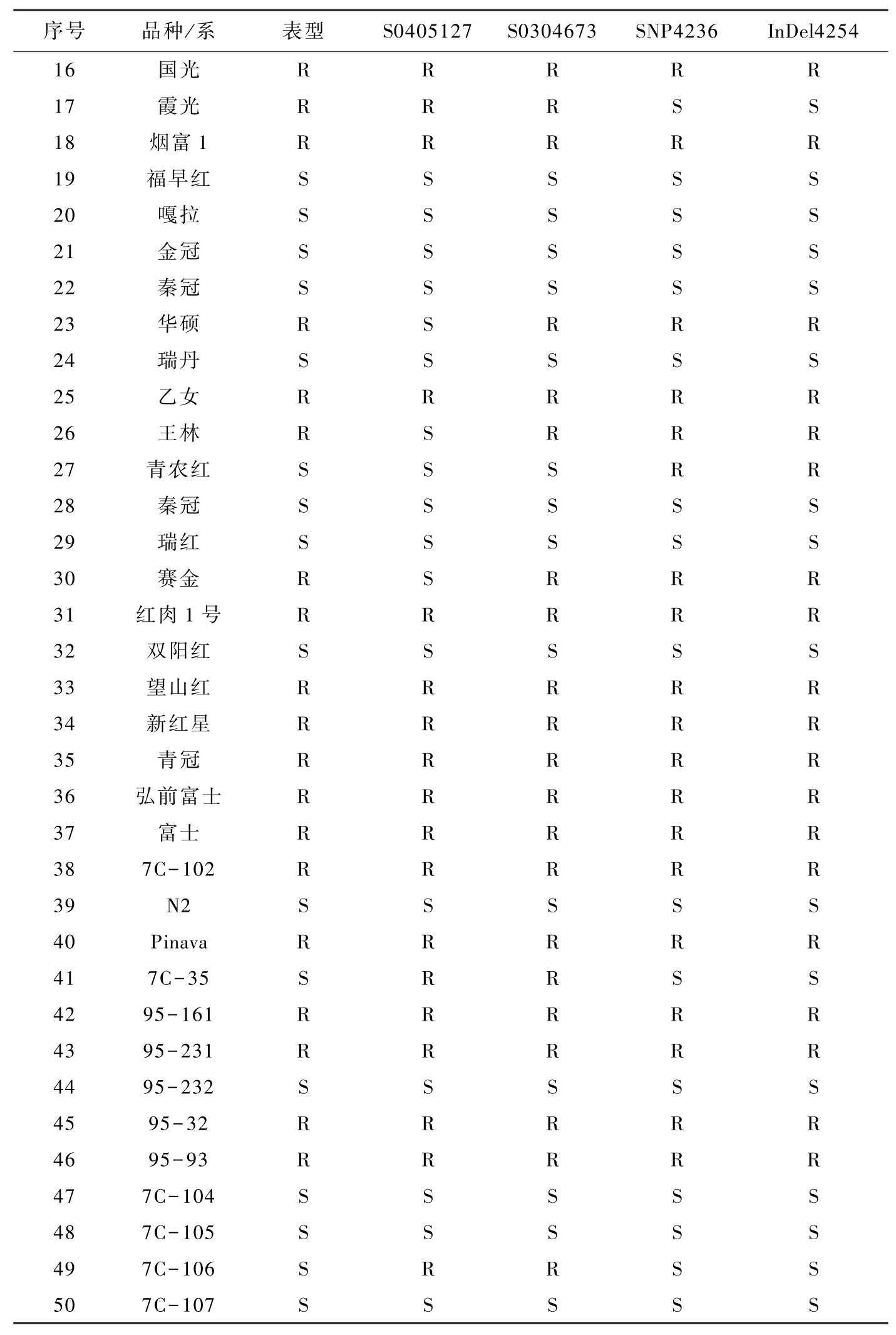
表5-3 四个分子标记对苹果栽培品种和品系的抗病性的鉴定结果(续)-1
第三节 讨论与小结
精细定位通常采用的方法是侧翼分子标记法,对于有参基因组植物来说,就是根据对目的基因的初步定位结果,选择位于目的基因两侧的分子标记之间的碱基,用于设计合适的分子标记。筛选出的与目的基因连锁的分子标记,通过鉴定更大的群体来确定发生交换的重组单株,最终找到与目的基因紧密连锁的分子标记,从而实现对目的基因的精细定位。精细定位是图位克隆策略中重要的一步,可以通过开发新的分子标记,整合原来已有的遗传图谱来进行图谱加密,以实现对目的基因的精细定位。
本研究利用全基因组重测序技术开发出的 SNP 及 InDel标记,结合
由于现有的金冠苹果基因组数据库中可能存在组装错误,所以在对定位区域内的基因进行分析中,扩大
在该区域内存在着4个编码WRKY转录因子的基因
进一步的基因功能研究及苹果与炭疽叶枯菌的分子互作机制研究将围绕着SNP 定位的5个候选基因及该区段内的相关基因展开。
随着分子生物学技术的快速发展,特别是以DNA多态性为基础的分子标记技术在苹果育种中的应用,大大提高了目标性状早期选择的效率,缩短了育种周期,加快了新品种选育的速率。Tartarini 等(2000) 报道了利用获得的与抗苹果黑星病的显性单基因
本实验利用四个紧密连锁的分子标记 S0405127、S0304673、SNP4236和 InDel4254对50个田间栽培品种和青岛农业大学选育出的优系进行了抗炭疽菌叶枯病的基因型鉴定,并结合其抗病的表型鉴定









