Screening and Analysis on T-DNA Insertional Pathogenicity Mutants of Magnaporthe grisea
Screening and Analysis on T-DNA Insertional Pathogenicity Mutants of Magnaporthe grisea
Keywords:
稻瘟病菌[
已知水稻与稻瘟病菌之间的特异性互作,符合Flor提出的“基因对基因”关系假说
稻瘟病菌基因组全序列测定已经完成
1 材料与方法
1.1 供试稻瘟菌株及水稻品种
稻瘟病菌菌株FJ95054B,是本研究的野生型对照菌株,由本实验室从福建田间单孢分离得到的菌株;供筛选的突变体菌株,是随机从本实验室已建成的稻瘟病菌菌株FJ95054B T-DNA插入突变体库中抽取的。
供接种筛选的水稻品种CO39近等基因系:C101LAC
1.2 突变体致病性的测定
稻瘟病菌菌株的活化、扩大培养及产孢方法参照王宝华等
1.3 突变体生长发育的观察
参考李宏宇方法
1.4 稻瘟病菌基因组DNA的提取及TAIL-PCR
稻瘟病菌基因组DNA的提取参照何月秋方法
将TAIL-PCR第2、3步扩增产物用0.5×TBE制备1.0%琼脂糖凝胶进行电泳检测,电泳电压为5 V/CM。电泳结束后,将凝胶放入含EB的0.5×TBE 中染色20~30min,用紫外透射反射仪观察并照相记录。
1.5 T-DNA侧翼序列的获得与生物信息学分析
TAIL-PCR扩增产物通过电泳分离后,回收目的片段,纯化后与pGEM-T Easy Vector(购自Promega公司)连接;连接产物通过热激转化到DH5α感受态细胞中,通过蓝白斑反应初筛转化子;采用碱裂解法少量提取转化子中的质粒DNA,通过PCR及酶切鉴定,挑选。
将含正确插入的转化子的克隆,送由上海鼎安生物科技有限公司测序。测序获得的序列去除T-DNA载体序列后,将剩余的序列与GenBank数据库(http://www.ncbi.nlm.nih.gov)、稻瘟菌基因组数据库(http://www.broad.mit.edu/annotation/fungi/magnaporthe/index.html)进行Blast比较分析及相关生物信息学分析,以推断T-DNA所插入的基因及其可能的功能。此外,应用SignalP 3.0 server(Http://www.cbs.dtu.dk/services/SignalP/)推断所获得的基因序列中是否含有信号肽(signal peptide,SNP);应用Pfam(http://www.sanger.ac.uk/Software/Pfam/)推测基因编码的蛋白可能的结构域;应用TMHMM-v 2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测基因编码的蛋白可能的跨膜结构域;应用Protcomp-v 6.0(http://sun1.softberry.com/berry.phtml)对基因编码的蛋白质进行亚细胞定位,该软件可将蛋白质按细胞核、质膜、胞外分泌、细胞质、线粒体、内质网等归属进行划分;应用TargetP-v 1.1(http://www.cbs.dtu.dk/services/TargetP/)进一步确定预测蛋白的亚细胞定位,其可将蛋白质的定位归属于线粒体、叶绿体、胞外分泌以及其他亚细胞定位。
2 结果与分析
2.1 致病性突变体的筛选
将随机选取的60个突变体与野生型菌株FJ95054B分别接种在国际水稻研究所提供的CO39近等基因系的6个水稻品种上,即C101LAC
菌株号IsolatesC101LACPi-1(t)C101A51Pi-2(t)C104PKTPi-3(t)C101PKTPi-4aC101TTP-4L-23Pi-4bCO39FJ95054B(WT)RRSRRS2t-2T940024501/++////T940084501/////—T940086501/////—
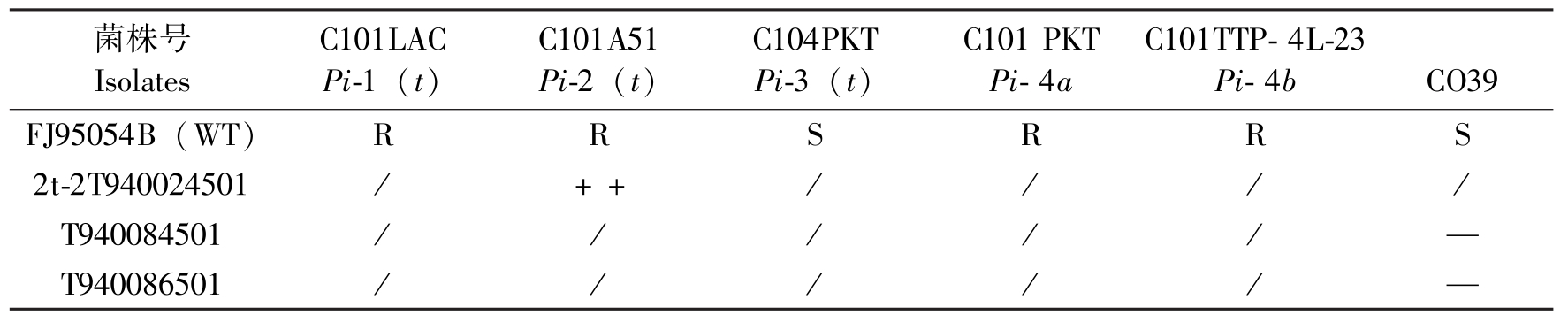
表1 稻瘟病菌T-DNA插入致病性突变体接种CO39近等基因系的发病情况*
Table 1 Virulence of T-DNA insertional pathogenicity mutants on CO39 NILs
2.2 致病性突变体的表型分析
稻瘟病菌侵染过程中的任何一个环节被破坏均会导致致病性减弱或丧失,因此,对致病
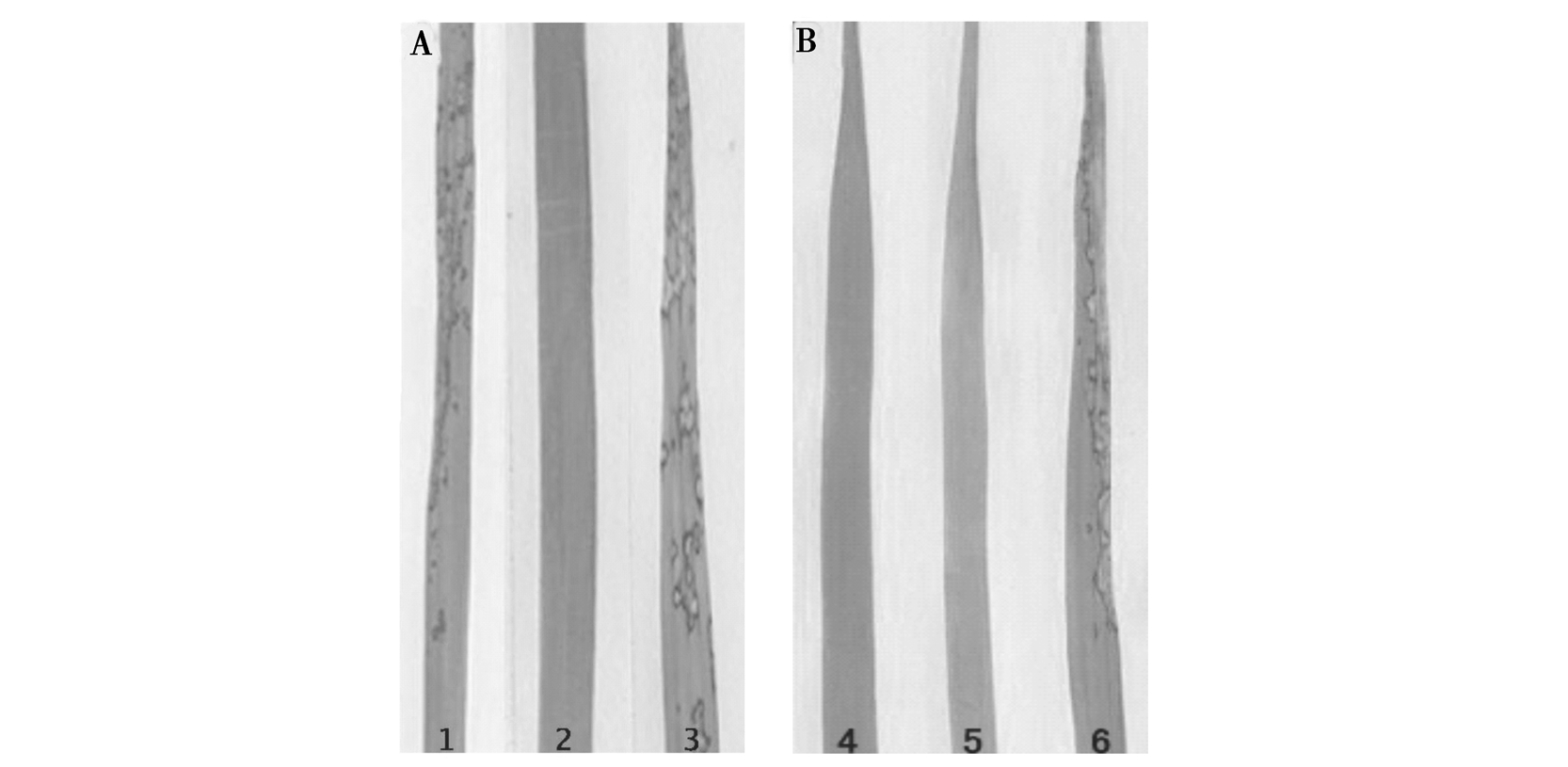
图1 稻瘟病菌致病性突变体的变化情况
Figure 1 Pathogenicity of mutants of M.grisea compared with wildtype FJ95054B
菌株Isolate菌落直径Colonydiameter(cm)产孢量Sporulation(105spores/cm2)8h孢子萌发率Percentageofsporegermination8hpi(%)16h附着孢形成率Percentageofappressoriumformation16hpi(%)95054B(WT)5.20±0.100.7499.0693.68T-9400845014.17±0.030.001287.9073.17T-9400865014.22±0.070.003383.8963.69
表2 稻瘟病菌致病性减弱突变体的生长发育变化
Table 2 Morphology and development of pathogenicity-decreased mutants of M.grisea
此外,在洋葱表皮侵入实验中,野生型FJ95054B侵入正常,能形成正常的附着胞以及侵染栓,并能观察到菌丝在洋葱细胞内广泛蔓延。相比之下,突变体T940084501的孢子能够形成附着胞,但随后并未形成侵染栓侵入,未发现菌丝在洋葱细胞内蔓延;而突变体T940086501的部分孢子能够形成附着胞,但同样未能形成侵染栓侵入,也未发现菌丝在洋葱细胞内蔓延,还有部分孢子甚至不会形成附着胞。这些都可能是其致病性减弱的原因。实验结果如图2所示。
图2 稻瘟病菌致病性减弱突变体的洋葱表皮侵入实验结果
Figure 2 The results of onion penetration assays of pathogenicity-decreased mutants of M.grisea
2.3 TAIL-PCR获得T-DNA插入的侧翼序列
使用右边界嵌套特异引物RB与随机简并引物AD7组合,扩增出了突变体2t-2 T940024501的T-DNA侧翼序列(如图3A所示);使用左边界嵌套特异引物LB与随机简并引物AD9组合,扩增出了突变体T940084501的T-DNA侧翼序列(如图3B所示)。突变体T940086501的T-DNA侧翼序列通过TAIL-PCR反应尚未获得。
2.4 相关生物信息学分析
通过在稻瘟菌基因组数据库进行的比对分析,结果显示突变体2t-2 T940024501 T-DNA侧翼序列中的117~591 bp区段与稻瘟病菌基因组supercontig5.183的776534~777008 bp同
图3 TAIL-PCR第二步和第三步产物的电泳图谱
Figure 3 Electrophoresis patterns of secondary and tertiary TAIL-PCR products(secondary and tertiary products of the same isolate are loaded side by side)
分别利用SignalP 3.0、Protcomp-v 6.0、TMHMM-v 2.0、TargetP-v 1.1以及Pfam对该基因编码的蛋白进行分析。首先通过Signal IP 3.0预测,并不包含信号肽结构;继而经TMHMM-v 2.0预测,不含跨膜结构;通过Protcomp-v 6.0和TargetP-v 1.1分析表明,含有胞外分泌信号,是一种胞外分泌蛋白,在胞内无定位。另外,Pfam分析结果表明,该基因可能具有某种糖基水解酶活性。
目前仅知该基因被T-DNA阻断后,突变体菌株对水稻品种
3 讨论
对稻瘟病菌无毒基因以及致病过程有关基因的研究一直是了解稻瘟病菌致病性及其变异机制乃至水稻与稻瘟病菌特异性互作机制的关键所在,而研究某一个具体基因的功能最便捷的方法就是对该基因的突变体进行分析,这也正是本研究筛选T-DNA插入所致致病性突变体的初衷所在。
获得致病性变异稳定的突变体是本研究的关键之一。本研究通过大量平行、重复实验初
本研究对所获得的基因的功能还只是做了初步的预测,为了明确其功能尚需要借助进一步的基因敲除和功能互补实验。另外,为了明确致病性变异是否为单基因作用所致,尚且需要对致病性突变体的后代群体进行接种实验和潮霉素抗性分析,通过检测其后代的表型分离比率加以验证。









