Analysis of Elongation Factor Gene and Ribosomal Protein Gene Sequence of Mulberry Dwarf Phytoplasma*
Analysis of Elongation Factor Gene and Ribosomal Protein Gene Sequence of Mulberry Dwarf Phytoplasma*
桑叶是家蚕的唯一饲料,桑树是蚕丝产业的物质基础。桑树生长在各种不同气候和地貌的生态环境,桑树萎缩病是蚕桑生产最严重的病害之一,主要有黄化型、萎缩型和花叶型3种类型,分布于中国、日本、韩国、格鲁吉亚、越南等蚕桑生产国,特别是中国和日本受害最为严重。桑树萎缩病在我国主要蚕桑区江浙、湖广及北方的山东、陕西等省均有分布,对蚕桑生产造成严重甚至毁灭性的危害,且防治比较困难。
本研究对山东省发生的症状为萎缩型的桑萎缩病,采用PCR技术对其16S rDNA、延伸因子和核糖体蛋白基因片段进行扩增和直接测序,并且与已知的植原体各组中代表的植原体序列进行同源性比较,确定了该分离物的亚组分类地位。现将研究结果报道如下。
1 材料和方法
1.1 材料
表现萎缩型症状的发病桑树材料采自山东省宁阳市。克隆载体pMD18-T、PCR产物回收试剂盒、限制性内切酶及其他酶类等分子生物学试剂产品均购自TaKaRa公司。
1.2 总DNA的提取
提取方法参照漆艳香等
1.3 引物设计和合成
植原体16S rDNA基因扩增采用Lee等
NamePrImersequenceTm(℃)ReferencesR16mF25′-CATGCAAGTCGAACGGA-3′60[3]R16mR15′-CTTAACCCCAATCATCGAC-3′60[3]fTufu5′-CCTGAAGAAAGAGAACGTGG-3′50[4]rTufu5′-CGCAAATAGAATTGAGGACG-3′50[4]rpF15′-GGACATAAGTTAGGTGAATTT-3′55[5]rpR15′-ACGATATTTAGTTCTTTTTGG-3′55[5]
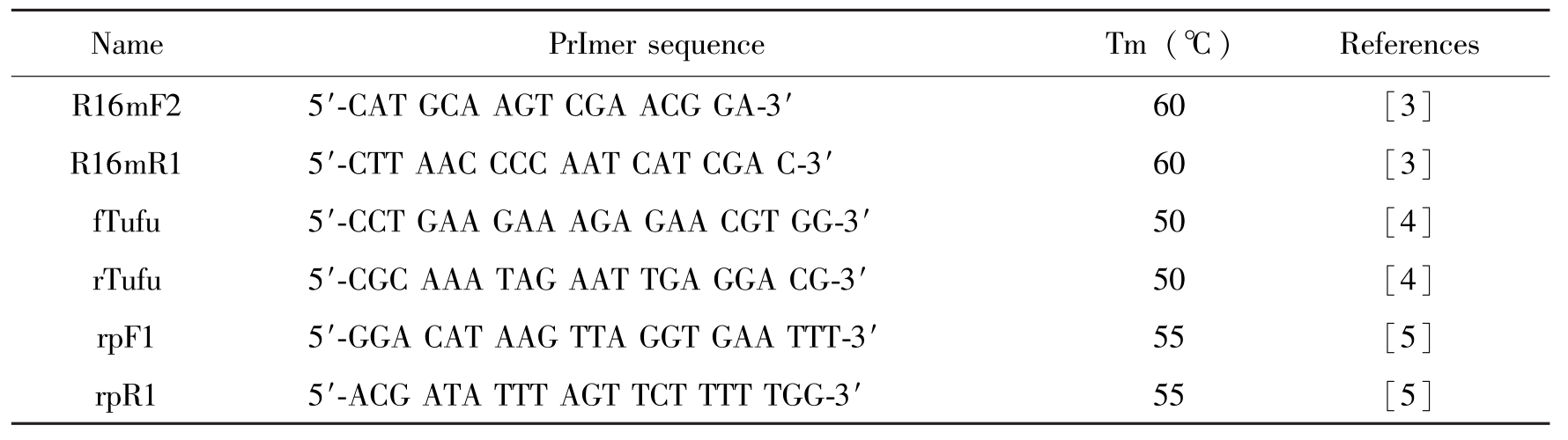
Table 1 Primers used in this research to amplify three gene
1.4 产物的克隆与序列分析
PCR产物采用1%琼脂糖凝胶电泳检测,并用PCR纯化试剂盒进行纯化回收。PCR产物回收后与pMD18-T连接,连接产物转化大肠杆菌DH5α感受态细胞,挑取筛选平板上的白色菌落培养,提取质粒,经PCR和酶切鉴定为阳性的重组质粒送上海英骏生物技术有限公司测序。将所得DNA 序列输入GenBank进行Blast检索,采用DNAMAN和MEGA3.1软件对所得到的核苷酸序列与GenBank中收录的相应基因的核苷酸序列进行比对分析,并构建系统进化树。
2 结果与分析
2.1 PCR扩增结果
以带有桑萎缩病原(Mulberry dwarf-Ningyang,MDNY)的发病桑树病组织总DNA为模板,16S rDNA引物经直接PCR扩增,得到长度为1.5kb的片段,延伸因子基因引物经PCR
2.2 基因序列测定及同源性比较分析
对含有目标外源片断的重组质粒进行序列测定,扩增得到16S rDNA基因片断为1431bp;延伸因子基因片断为842bp,共编码280个氨基酸;核糖体蛋白基因片断为1249bp,DNAMAN软件分析表明该序列包括部分rps19和全部rp122和rps3基因。
将MDNY的16S rDNA序列与GenBank中22个植原体分离物16S rDNA的核苷酸序列进行比对和构建系统进化树,该分离物与植原体的16S rI组处于同一个分支,与16S rI-B和16S rI-D亚组同源性最高。
该分离物MDNY的延伸因子基因序列与GenBank中植原体16S rI组的7个亚组分离物延伸因子基因的核苷酸序列进行比对和构建系统进化树(Figure 1)。从系统进化树中可以看出,该分离物MDNY与16S rI-B和16S rI-D亚组处于同一分支,同源性最高,与16S rDNA的结果一致。

Figure 1 Phylogenetic tree of MDNY based on the elongation factor gene sequences using neighbor-joining in MEGA3.1
该分离物MDNY的核糖体蛋白基因(rp)序列与GenBank中植原体16S rI组的6个亚组分离物核糖体蛋白基因的核苷酸序列进行比对并构建系统发育树(Figure 2),从系统进化树中可以看出,该分离物与16S rI-D 的Paulownia witches-broom处于同一分支,同源性最高,确定了该分离物的亚组分类地位。

Figure 2 Phylogenetic tree of MDNY based on the rIbosomal protein gene sequences using neighbor-joining in MEGA3.1
3 讨论
夏志松等对桑黄化性萎缩病病原体16S rRNA基因序列进行了分析
20世纪80年代至今,由于分子生物学技术,特别是分子克隆和PCR技术的应用大大加快了包括植原体在内的细菌系统学研究进展。目前植原体的分类16S rDNA是一种非常有效的手段,但对于16S rDNA类群内的进一步划分,用16S rDNA类群作为分类标准显得过于保守,延伸因子和核蛋白基因序列可以作为系统学研究的依据。Schneider等
本试验以16S rDNA基因,延伸因子基因及核糖体蛋白基因作为分类依据,在亚组水平明确了桑树萎缩病的分类地位,为今后研究桑树萎缩病植原体的来源、进化关系及其致病的分子机理提供了理论依据。









